Trồng dâu nuôi tằm, lấy tơ dệt lụa là một trong những nghề có thể giúp xoá đói giảm nghèo, nâng cao đời sống cho người lao động ở vùng nông thôn, vùng sâu, vùng xa. Nhu cầu về các sản phẩm tự nhiên như tơ tằm ngày càng tăng, vì vậy đòi hỏi ngành dâu tằm phải có các biện pháp để nâng cao năng suất và chất lượng sản phẩm. Tuy nhiên cho đến nay, vấn đề lựa chọn giống tằm có năng suất cao và sức chống chịu tốt vẫn còn hạn chế do việc chọn tạo giống tằm trong nước chủ yếu vẫn thực hiện theo phương pháp truyền thống (đánh giá giống, lựa chọn bố mẹ trong cặp lai mới chỉ dựa vào kiểu hình và đặc điểm sinh học nên tốn nhiều thời gian mà kết quả mang lại chưa như mong đợi).
Những năm gần đây, sử dụng chỉ thị phân tử DNA để phân tích di truyền các giống vật nuôi, cây trồng đã trở thành một hướng nghiên cứu quan trọng trong lĩnh vực công nghệ sinh học phục vụ đời sống con người. Các thông tin di truyền phân tử đã trợ giúp cho công tác giống để phát triển tập đoàn dòng thuần, tìm hiểu ưu thế lai, xác định các cặp lai ưu tú. Đánh giá đa dạng di truyền của một số giống tằm hiện có tại Việt Nam là vấn đề quan trọng đối với sự thành công trong việc lựa chọn, ghép đôi, tạo giống ở tằm. Việc đánh giá đa dạng di truyền cung cấp thông tin về một số khác biệt giữa các giống tằm, là nền tảng cho các chương trình nhân giống có mục tiêu cụ thể. Sự đa dạng di truyền của tằm (B. mori) bắt nguồn từ sự lai tạo giữa các giống có nguồn gốc địa lý khác nhau, chủ yếu là các giống đến từ Nhật Bản, Trung Quốc, châu Âu và Ấn Độ. Việc thuần hóa và chọn lọc để tạo ra các giống tằm có năng suất tơ lụa cao đã làm tăng tính đồng nhất di truyền dẫn đến mất khả năng chống chịu với điều kiện thời tiết biến đổi và khả năng đối phó với dịch bệnh. Do đó, duy trì sự đa dạng di truyền là một chiến lược cơ bản trong quản lý lâu dài để bảo tồn và cải thiện di truyền của tằm (Bindroo & Manthira Moorthy, 2014). Cùng với việc bảo tồn nguồn gen qua việc tuyển chọn, nuôi dưỡng, nhân giống, việc đánh giá đa dạng di truyền sử dụng marker phân tử có thể như một hướng dẫn ban đầu để xác định nguồn gen độc đáo và có giá trị phục vụ cho việc bảo tồn các nguồn gen quý một cách chính xác (Nagaraju & Goldsmith, 2002).
Trình tự nucleotide của gen ty thể (mtDNA) đã được nhiều nghiên cứu sử dụng trong đánh giá đa dạng di truyền giữa các loài và trong cùng một loài (Li & cs. 2005; Zanatta & cs., 2009; Yukuhiro & cs., 2011; Fassina & cs., 2014; Singh & cs. 2017; Vimala & cs., 2020; Kim & cs., 2019; Zhang & cs. 2019; Kim & cs., 2021; Alcudia-Catalma & cs., 2021). Trình tự nucleotide gen COI của mtDNA cũng đã được nhiều nghiên cứu sử dụng để đánh giá đa dạng di truyền giữa các giống tằm trên thế giới (Kim Ik Soo & cs., 2000; Yukuhiro & cs., 2011; Fassina & cs., 2014; Vimala & cs., 2020). Các ứng dụng của trình tự nucleotide gen ty thể trong đánh giá đa dạng di truyền giữa các chủng và giống tằm trên thế giới đã được nghiên cứu, tuy nhiên tại Việt Nam chưa có nghiên cứu nào sử dụng trình tự nuclotide gen COI để đánh giá đa dạng di truyền. Do đó, nhóm tác giả đã thực hiện nghiên cứu “Đánh giá đa dạng gen COI ở tằm dâu (Bombyx mori)” với nguồn vật liệu là 14 giống tằm bao gồm 9 giống tằm lưỡng hệ kén trắng có nguồn gốc Ấn Độ, Hàn Quốc, Trung Quốc, Việt Nam và 5 giống tằm đa hệ kén vàng bản địa Việt Nam.
Kết quả
Khuếch đại gen COI ở tằm
Kết quả khuếch đại gen COI, cho thấy đoạn gen đã được nhân lên đặc hiệu với 1 băng DNA sáng rõ, kích thước phân tử tương ứng khoảng 658 bp, phù hợp với kích thước theo tính toán lý thuyết (Hình 1).
|
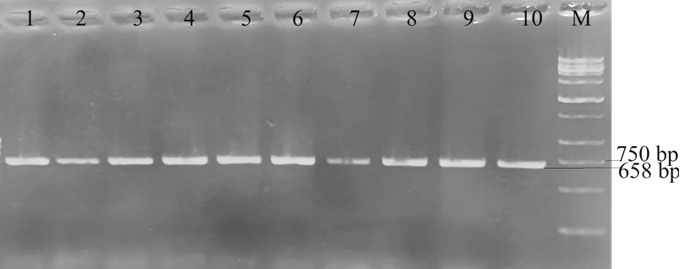
Hình 1. Hình đại diện sản phẩm khuếch đại gen COI ở tằm
|
|
Sản phẩm PCR sau khi được kiểm tra chất lượng, được tinh sạch và tiến hành đọc trình tự gen trực tiếp.
Phân tích trình tự gen COI các giống tằm
Trình tự nucleotide gen COI từ 14 giống tằm (9 giống lưỡng hệ kén trắng và 5 giống bản địa đa hệ kén vàng) đã được gửi vào GenBank, mã số từ OR610743 đến OR610756. Kết quả so sánh trình tự nucleotide gen COI của 14 giống tằm với trình tự AB737913.1 (trình tự nucleotide gen COI của tằm hoang dã Trung Quốc, nhiều nghiên cứu trước đây cho thấy rằng, B. mori có nguồn gốc từ tằm hoang dã Trung Quốc) cho thấy, có 17 vị trí đa hình nucleotide, tất cả các vị trí đa hình đều thay thế nucleotide, không có vị trí đa hình thêm hay mất nucleotide. Sự thay thế nucleotide xuất hiện chủ yếu từ T thành C hoặc ngược lại từ C thành T (13/17 vị trí đa hình) (Hình 2), kết quả này đồng thuận với nghiên cứu của Kim Ik Soo & cs. (2000). Còn khi so sánh trình tự nucleotide gen COI giữa 14 giống tằm nghiên cứu chỉ xuất hiện một vị trí thay thế nucleotide từ A thành G (vị trí 179). Thành phần nucleotide của tất cả các haplotype là 33,14% A; 37,00% T; 15,38% C; 14,48% G; 70,14% A+T và 29,86% G+C. Kết quả này tương đồng với các nghiên cứu trước đây khi giải trình tự nucleotide gen COI: tỷ lệ nucleotide A+T chiếm 70,2%, còn C+G đạt 29,8% (Vimala & cs., 2020), tỷ lệ A+T đạt (70,3%), C+G (29,7%) (Fassina & cs., 2014).
|
 Hình 2. So sánh tương đồng trình tự nucleotide gen COI (658bp) giữa 14 giống tằm và trình tự nucleotide trên Genbank (AB737913.1) Hình 2. So sánh tương đồng trình tự nucleotide gen COI (658bp) giữa 14 giống tằm và trình tự nucleotide trên Genbank (AB737913.1)
|
|
Sử dụng phần mềm DnaSP để xác định haplotype trên 658 nucleotide gen COI của 14 giống tằm. Kết quả cho thấy các giống tằm nghiên cứu được phân bố thành 2 haplotype, trong đó giống tằm Vàng Bảo Lộc nằm ở haplotype 1, còn 9 giống tằm lưỡng hệ kén trắng và 4 giống tằm bản địa Việt Nam mặc dù được thu thập từ nhiều nước khác nhau (Ấn Độ, Trung Quốc, Hàn Quốc và Việt Nam) nhưng đều tập trung vào haplotype 1 (Hình 3). Kết quả này khá tương đồng với nghiên cứu của Fassina & cs. (2014) khi nghiên cứu trên các giống tằm từ Trung Quốc, Ấn Độ, Nhật Bản và các giống lai, nhưng không tìm thấy sự khác biệt đáng kể nào giữa các giống tằm. Kết quả này tương đồng với kết quả nghiên cứu của Nguyễn Thị Thanh Bình & Đặng Đình Đàn (2008), khi sử dụng chỉ thị phân tử RAPD để nghiên cứu sự trùng lặp giữa các giống tằm bản địa, và kết luận rằng giống Ré Vàng Thái Bình và Hoàng Liên Sơn có thể bắt nguồn từ một giống nhưng có hai tên gọi khác nhau.
|
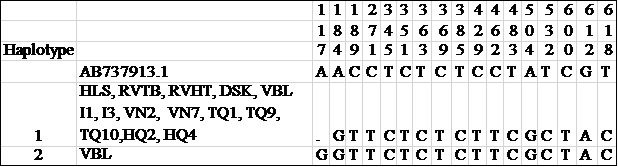
Hình 3. Sự phân bố của các giống tằm trong các haplotype
|
|
Sử dụng phần mềm DNAsp để tính các chỉ số Hd, Pi, kết quả cho thấy, chỉ số đa dạng haplotype (Hd) gen COI ty thể của 14 giống tằm nghiên cứu đạt (Hd: 0,1429), kết quả này cho thấy các giống tằm ở Việt Nam ít đa dạng hơn so với các giống tằm ở Nhật Bản (Hd: 0,4140), Trung Quốc (Hd: 0,2914), Châu Âu (Hd: 0,2540) và chủng Moltinism (Hd:0,4238) và thấp hơn rất nhiều so với Bombyx mandarina (Hd: 0,8730). Chỉ số đa dạng nucleotide (Pi) của 14 giống tằm trong nghiên cứu đạt 0,00022 thấp hơn hơn các giống tằm ở Nhật Bản (Pi: 0,00059), Trung Quốc (Pi: 0,00048), chủng Moltinism (Pi: 0,00079), Bombyx mandarina (Pi: 0,00224), châu Âu (Pi: 0,00036) (Yukuhiro & cs., 2011).
Cây phân loại di truyền giữa các giống tằm nghiên cứu và các giống tằm trên thế giới
Cây phân loại di truyền được xây dựng từ 14 giống tằm nghiên cứu, 19 chủng tằm phân bố trên 8 nhánh theo Yukuhiro & cs. (2011) và 6 chủng tằm hoang dã trên Genbank, kết quả cho thấy rằng 14 giống tằm nghiên cứu phân bố trong 2 nhánh (Hình 4.). Trong đó 13/14 giống tập trung ở nhánh 2, đây là nhánh phân bố phổ biến của các chủng tằm Trung Quốc (42/50 giống), chủng tằm châu Âu (24/28) và các chủng Moltinism (16/21). Còn giống tằm Vàng Bảo Lộc phân bố trong nhánh 1, đây là nhánh phân bố phổ biến của các chủng tằm Nhật Bản (35/48 chủng tằm Nhật Bản phân bố ở nhánh 1), và một số ít chủng tằm Trung Quốc, châu Âu và Moltinism cũng nằm trong nhánh này (Yukuhiro & cs., 2011). Nghiên cứu của Fassina & cs. (2014) cho thấy đa phần các chủng tằm nghiên cứu tập trung vào nhánh 1.
|
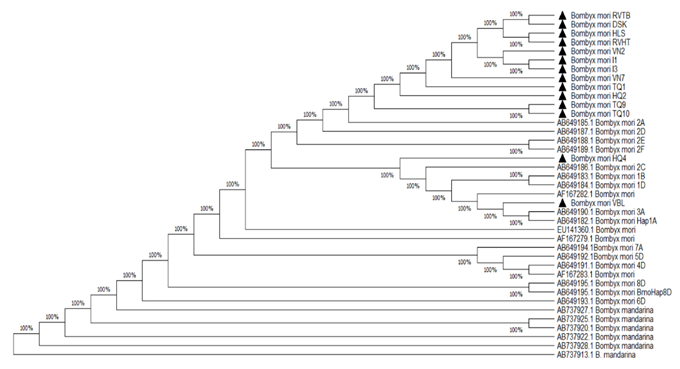 Hình 4. Cây phân loại di truyền của 14 giống tằm nghiên cứu và các giống tằm trên thế giới Hình 4. Cây phân loại di truyền của 14 giống tằm nghiên cứu và các giống tằm trên thế giới
(Các giống tằm nghiên cứu được đánh dấu tam giác, màu đen)
|
|
Kết luận
Tại nghiên cứu này, nhóm nghiên cứu đã phân tích 658 bp trình tự nucleotide gen COI của 14 giống tằm cho thấy, tỷ lệ nucleotide A+T đạt 70,14%, G+C đạt 29,86%. Có 17 đa hình nucleotide khi so sánh 14 giống tằm nghiên cứu với tằm hoang dại Trung Quốc có mã số Genbank AB737913.1. Chỉ xuất hiện một vị trí đa hình nucleotide đơn khi so sánh 14 giống tằm với nhau. 14 giống tằm nghiên cứu nằm trên 2 haplotype và phân bố trên 2 nhánh. Kết quả này cho thấy mức độ đa hình trình tự nucleotide gen COI thấp khi so giữa các giống tằm.
Kiến nghị
Trình tự 658 nucleotide gen COI chưa bộc lộ hết tính đa dạng của các giống tằm, vì vậy nghiên cứu tiếp theo có thể sử dụng các trình tự nucleotide khác như Cytb, Dloop, hoặc toàn bộ trình tự gen ty thể để có cái nhìn rõ nét hơn về các giống tằm nuôi tại Việt Nam.
VIỆN NGHIÊN CỨU TĂNG TRƯỞNG XANH
Adress: Học viện Nông nghiệp Việt Nam, Trâu Quỳ, Gia Lâm, Hà Nội.
Tel: 024 66583219 (Office); Hotline: 098 2332755; Fax: 024.38361283
Email: agg@vnua.edu.vn Web: agg.vnua.edu.vn